第一代Sanger测序法,由Sanger等在1997年提出,基本原理是通过双氧核糖核苷酸进行延伸反应,生成相互独立的若干组带放射性标记的寡核苷酸,再通过凝胶电泳使它们分开,最后通过放射显影读出待测DNA上的核酸序列。
第二代(Next-generation sequencing,NGS)高通量测序法,引入可逆终止末端,并对其荧光标记,从而实现边合成边测序。优点是高通量,可定量,成本低廉,但是读长较短,一般不超过500bp。
第三代测序技术即单分子测序技术,包括单分子实时(Single Molecule Real-time,SMRT)测序技术和单分子纳米孔(Nanopore)测序技术,优点为超长读长且无需扩增。
虽然第三代测序已经商用了,但目前主流测序技术还是NGS,以下从其测序过程对其简单介绍。
一、文库构建
1、基因组文库构建
① 通过物理性(超声波法、喷雾法或水动力剪切等)机械打碎和酶消化切割等手段将提取到的基因组DNA随机打断。
② 打断的DNA片段末端可能带有5’—凸出和3’—凸出,且其末端不一定存在磷酸基团或者羟基基团。因此,需要使用Taq聚合酶补齐不平的末端,同时使用Klenow 酶在两个末端添加碱基A。此过程即为末端修复,修复后的即可链接测序接头。
③ 测序接头是已知的用于测序识别的序列,包括有与固定在流动槽中寡核苷酸序列互补的序列(P5/P7)和簇生成引物序列(Read SP)以及标签序列(Index),添加完接头序列的DNA片段集合即为所建基因组文库。
④ 建库过程中含有聚合酶、连接酶等以及其他各种杂质,需要对其进行磁珠纯化,纯化过程中同时进行片段选择。纯化后的片段两端是不互补的Y形结构,不能直接进行测序,所以还要进一步用与接头互补的引物来PCR扩增。扩增完,进一步磁珠纯化,洗去杂质。
2、转录组文库构建
转录组是指特定细胞或组织中全部转录产物,包括信使RNA(Messenger RNA,mRNA)、核糖体RNA(Ribosomal RNA,rRNA)、转运RNA(Transfer,tRNA)以及其他非编码RNA(noncoding RNA,nc RNA)。不同类型的RNA文库制备方法各不相同,主要过程有:
① 目标RNA富集,方法包括靶向捕获目标RNA和消除rRNA。前者通常使用Oligo-dT与Poly(A)杂交的特性设计探针,从而实现总RNA中捕获到聚腺苷酸化RNA;后者方法较多,包括有Ribo-Zero-seq法、RNA消化酶法以及根据rRNA特性来消除rRNA的方法等。
② 通过化学方法或者酶消化的方法打断富集到的RNA,或者将RNA反转录成cDNA,运用DNA片段化方法打断cDNA。
③ 连接接头。cDNA测序接头连接方法与DNA建库方法一致;小RNA长度20—35nt且5’端有磷酸基团、3’端有羟基基团,因此不需要特殊修饰即可与接头连接。
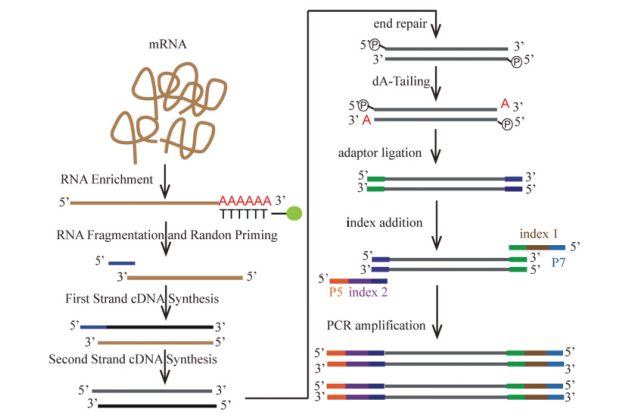
NEBNext Ultra II RNA 试剂盒( New England Biolabs) 文库制备流程
二、成簇Cluster Generation
利用有单链引物的流动槽将DNA分子片段固定在流动槽上扩增,形成单克隆DNA簇。
① 文库片段(P5/P7)与芯片表面引物互补配对,被固定后进行DNA复制。复制完后解链,将文库片段洗去,留在流动槽表面的即为文库模板互补的DNA链。
② 留下的互补链与原先模板链连接的引物结合,形成单链桥,再在聚合酶的参与下生成互补链,最终形成双链桥。
③ 双链桥再次变性后形成单链,形成的单链又分别与自己配对的引物结合,重复这个循环,形成散布在芯片上的DNA簇。
④ 进一步再切断洗去反向链(与模板链一致),仅留下正向链,且封锁3’端防止重新形成单链桥。
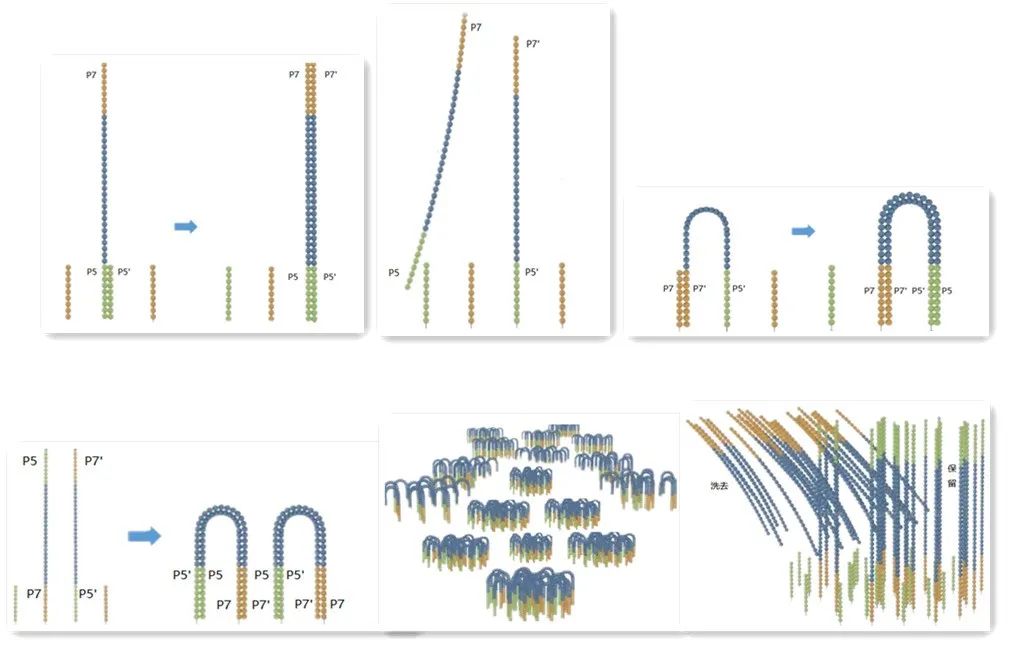
成簇Cluster Generation
三、测序Sequencing
向已经处理过的流动槽中添加改造过的DNA聚合酶和带有荧光标记的可逆终止dNTP(3’-OH链接叠氮基团,在延伸时被阻止连接下一个dNTP),统计每轮收集到的荧光信号结果,就可以得知每个模板DNA片段的序列。由于测序仪每次测序时的通量比较大,所以每次测得的序列可能不止一个样本,为了做区分,在建文库时在接头序列加入不同Index(或Barcode)区分来源。
现在主流测序方法为双末端(Paired-end)测序,因为可以增长测序长度且方便分析结构变异。一般是洗掉前面复制好的合成片段,单链继续在流动槽表面形成“桥式连接”。NaOH使双链变性为单链,并洗去已经测序完成的P7上DNA,留下P5’链。加入簇生成引物Read2,从相反方向进行另一端序列读取。
四、测序数据
以上则为二代测序的整个过程,不同的平台会存在一些差异,但总体过程类似。测序完成后,基于文库构建时添加的Index分类来自不同样本的序列,这些序列再与参考基因组匹配后,得到完整序列。